

  |
|
|
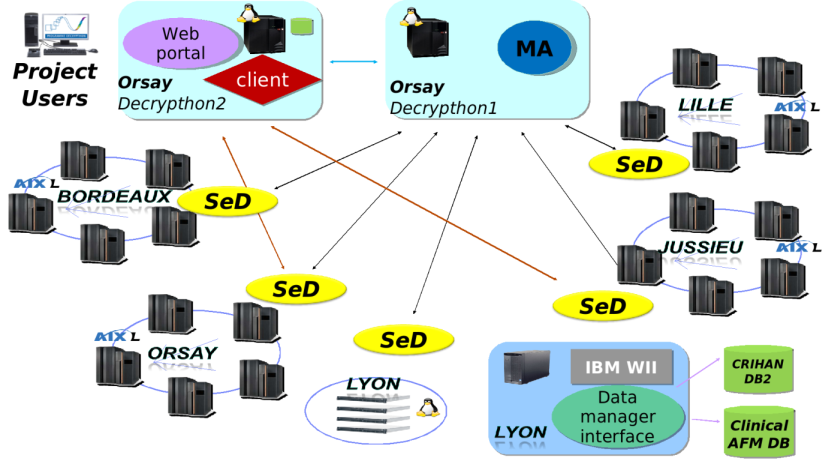
Le projet Décrypthon est le fruit de la collaboration entre le CNRS, l'Association Française contre les Myopathies AFM, et IBM. Il vise à fournir des ressources de calcul et des espaces de stockage mémoire aux chercheurs en bio-informatique. Ces ressources, situées dans des universités, constituent une grille de six sites : Bordeaux, Jussieu, Lille, Lyon, Orsay et Rouen, reliés par le réseau RENATER. La grille universitaire Décrypthon est pilotée par l'intergiciel DIET, qui permet le déploiement d'applications client-serveur sur un grand nombre de machines. |
 |
|
La grille Décrypthon s'articule autour de l'intergiciel DIET, d'un portail web qui permet l'accès aux ressources, et des gestionnaires de ressources locaux à chaque site. DIET est déployé sur tous les sites : des "Server Daemons" (SeD) sont lancés sur leurs frontales et sont capables de soumettre des jobs aux gestionnaires de batch locaux (OAR sur le cluster Linux de Lyon, LoadLeveler sur les sites AIX). Le serveur linux Decrypthon2 héberge les clients DIET, contrôlés par une interface web, et qui communiquent avec le master agent sur Decrypthon1 et avec les SeDs. |
| Du point de vue des utilisateurs des projets, une interface web dédiée à leur application est développée, leur permettant de soumettre des jobs et de télécharger leurs résultats. |
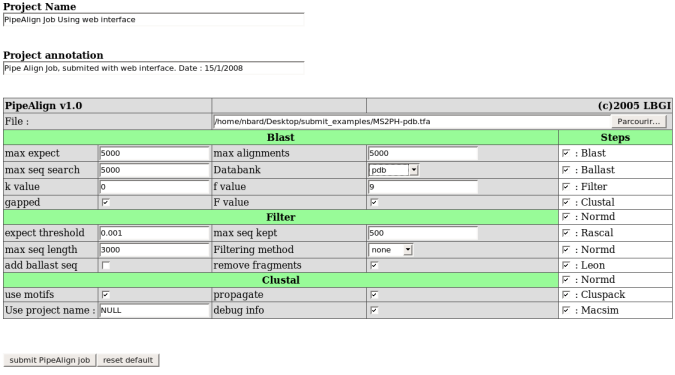
Le projet MS2PH : Olivier Poch (Illkirch, IGBMC / CNRS / Inserm) et Gilbert Deléage (Lyon, IBCP/CNRS) " Mutations structurales avec les conséquences sur le phénotype des pathologies humaines " |
|
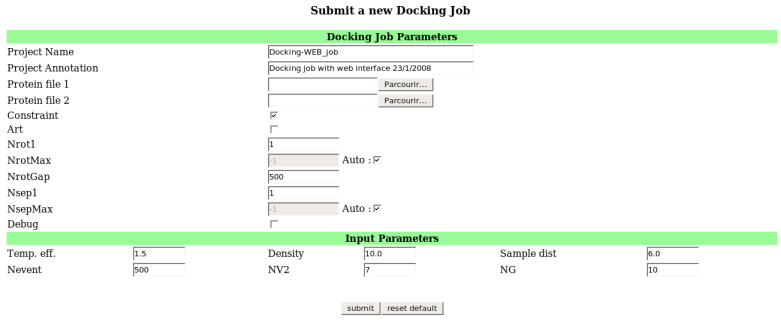
Le projet Docking : Alessandra Carbone de l'université Pierre et Marie Curie, Paris (Inserm U511 - Immunologie cellulaire et moléculaire des infections parasitaires - Génomique analytique) |
|
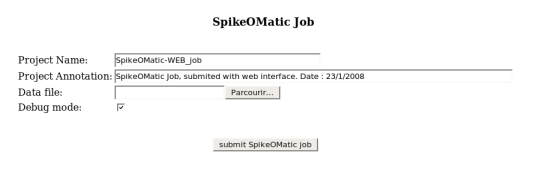
Le projet SpikeOMatic : Projet coordonné par Christophe Pouzat de l'université René Descartes, Paris V (CNRS UMR 8118 - Laboratoire de physiologie cérébrale) |
|
Telechargement des résultats : |
|