|
Personnes
|
|
Voici une courte liste des personnes avec qui j'ai collaboré afin de comprendre et mettre en place l'application au sein de la grille Décrypthon.
- Sophie SACQUIN-MORA (IBPC)
- Jean-Luc LAMOTTE (LIP6)
- Alessandra CARBONE
- Richard LAVERY
- Équipe IBM
WorldCommunityGrid
(Viktors BERSTIS, Bill BOVERMANN ...)
|
|
|
Présentation
|
|
|
projet HCMD (Help Cure Muscular Dystrophy) : Il s'agit d'étudier la modélisation des interactions protéine-protéine de protéines dont la structure tridimensionnelle (structure 3D) est connue, en se concentrant sur les protéines qui jouent un rôle dans les maladies neuromusculaires. Les informations obtenues seront rassemblées dans une base de données pour aider les chercheurs à concevoir les molécules susceptibles d'inhiber ou de favoriser la liaison de molécules particulières. L'objectif est de pouvoir trouver des traitements contre les myopathies et d'autres pathologies neuromusculaires.
L'approche proposée dans ce projet combine l'utilisation d'informations évolutionnistes (comment l'évolution a modifié les protéines pour améliorer leurs fonctions) et la modélisation moléculaire (détermination par calculs informatiques de la position relative de deux partenaires qui interagissent entre eux) pour identifier des interactions potentielles entre deux protéines.
Pour des structures complexes comme les protéines (les plus petites sont composées de centaines d'atomes), la modélisation par des moyens informatiques des interactions protéine-protéine demande beaucoup de temps.Ainsi, pour les 168 protéines sélectionnées initialement dans le projet, le temps de calcul nécessaire pour un ordinateur PC à 2 GHz a été estimé à plus de 14 siècles.
Le projet HCMD, dirigé par le Dr.
Alessandra Carbone
de l'Université "Pierre et Marie Curie" de Paris, est associé à des équipes spécialisées utilisant les grilles de calcul distribué (Jean-Marie Chesnaux du CNRS : Centre National français de la recherche scientifique), modélisation moléculaire (Richard Lavery, du CNRS), et analyse génétique des myopathies (Pascale Guicheney de l'INSERM, l'institut public français de recherche sur la santé).
Devant la quantité énorme de calcul demandé par ce projet, la grille Décrypthon composé de super-calculateurs n'est pas adapté; Nous nous sommes tourner
vers l'équipe du WorldCommunityGrid qui est en quelque sort l'équivalent de programme Décrypthon avec cependant une différence majeure : Ils utilisent une grille
de calcul volontaire : Elle se compose des ordinateurs des internautes qui désirent aider la recherche scientifique en offrant la puissance de leur machine lorsque celle-ci n'est pas utilisée.
- Biologie computationnelle : équipe d'
Alessandra Carbone
, Génomique analytique, Laboratoire de l'INSERM U511, Faculté de médecine, Université "Pierre et Marie Curie", Paris.
- Grille de calcul distribué : équipe de
Jean-Marie Chesneaux
, Laboratoire d'informatique de Paris 6 (LIP6), Laboratoire du CNRS UMR 7606, Université "Pierre et Marie Curie", Paris.
- Myopathies : équipe de
Pascale Guicheney
, Laboratoire de l'INSERM U582, Institut de la myologie, Hôpital de la "Pitié-Salpêtrière", Paris.
- Modélisation moléculaire : équipe de
Richard Lavery
, Laboratoire de biochimie théorique, Laboratoire du CNRS UPR9080, "Institut de Biologie Physico-Chimique", Paris.
|

|
|
|
|
Lancement du projet sur WorldCommunityGrid
|
|
|
Depuis le 19 Décembre 2006, le projet HCMD est lancé sur la grille de calcul volontaire WorldCommunityGrid.
Ce projet se composera de deux phases :
- Phase I : 168 protéines appartenant à une base de données de référence sont appariées 2 à 2. Les résultats de cette première étape
permettront par la suite aux chercheurs de réduire l'espace d'exploration aux seules zones pertinentes.
- Phase II : à partir des résultats de la phase I, les algorithmes permettant de faire un tri sur les zones pertinentes sera validé sur un
ensemble de 4000 protéines
|
|
|
|
|
Avancement
|
|
| Calculées | Total |
| nombre de protéines | 168
( 100 %) | 168 |
| temps CPU estimé | 14 s 88 a 237 j 19 h 45 min 54 s
( 100 %) | 14 c 88 y 237 d 19 h 45 min 54 s |
| quantité de données | 123 Go
( 100 %) | 123 Go |
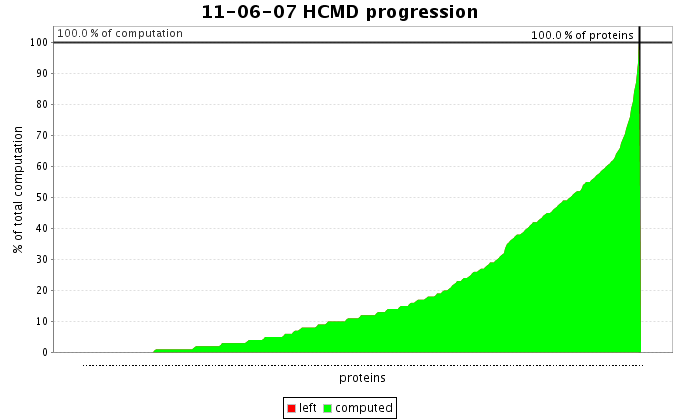
Le graphique ci-dessus représente l'avancement du projet HCMD.
- L'axe des ordonnées représente le pourcentage cumulé du temps CPU estimé.
- L'axe des abscisses représente le pourcentage cumulé des protéines.
- La partie verte représente les protéines qui ont été traité par les internautes participant au
WorldCommunityGrid
- La partie rouge représente les protéines en attentes d'être traitées ( ou en cours de calcul)
La phase I du projet HCMD consiste à obtenir la cartographie d'un ensemble de 168 protéines
de références.
Le temps CPU pour l'ensemble des protéines est estimé à plus de 14 siècles sur un processeur AMD Opteron(tm) Processor 246 (2Ghz).
j'ai obtenu cette estimation en effectuant une partie des calculs sur 600 machines
de la grille
GRID'5000
.
. Ce qui a représenté quelques 28224 jobs.
Comme vous pouvez le voir sur le graphique la courbe a une forme exponentielle. Nous avons commencé par les protéines qui demandent le moins
de calcul (la quantité de calcul est une fonction croissante de la taille de la protéine). Et l'ordre d'envoi des calculs respect l'ordre croissant des protéines.
Nous terminerons donc par les plus gourmandes en calcul.
Vous pouvez trouver d'autres informations sur le projet HCMD en visitant le site
décrypthon
Vous trouverez aussi des statistiques sur le projet HCMD sur le site du
WorldCommunityGrid
Vous remarquerez certainement que les statistiques issues du WorldCommunityGrid montrent que le projet a déjà consommé plus de 80 siècles de temps CPU. Cette
quantité est bien supérieure à la quantité estimée à partir des machines de la grille grid'5000. Quelques éléments de réponse peuvent être apportés :
- Les processeurs de la grille WorldCommunityGrid ne sont pas équivalent aux processeurs de la grille grid'5000.
- La grande hétérogénéité des machines participant à la grille WorldCommunityGrid implique de devoir utiliser un binaire compatible avec tout les types
de processeurs. Nous ne pouvons pas tirer partie des même optimisations que nous avions utilisé pour faire l'estimation du temps de calcul sur la grille Grid'5000
- Certains résultats sont calculés plusieurs fois; soit parce qu'ils sont erronés, soit parce qu'ils dépassent le délai d'attente que l'on s'est fixé pour obteinir les résultats
- Les processeurs des internautes ne sont pas dédiés aux jobs envoyés par le WorldCommuntyGrid, ils peuvent être partagés avec d'autres applications du système hôte.
Toutes ces raisons, montrent qu'il existe un facteur 6 entre le temps CPU estimé sur la grille Grid'5000 et le temps réel d'exécution sur les
machines du WorldCommunityGrid. Cependant nous tentons de réduire ce facteur en réduisant le nombre de calcul redondant. De plus il ne faut pas oublier
que le temps CPU total que j'ai estimé reste une estimation avec certaines hypothèses.
Cependant le fait que le WordlCommunityGrid comporte plus de 300 000 membres et plus de 650 000 machines
pouvant exécuter du code permet d'être optimiste sur la fin du projet.
Si vous ne participez pas encore à cette formidable aventure, je vous conseille vivement d'aller vous inscrire à partir du
site
décrypthon
, ou par le site
WorldCommunityGrid.
|
|
|